El Metotrexate (MTX), un análogo estructural del ácido fólico, es un fármaco muy eficaz en el tratamiento de enfermedades oncológicas y autoinmunes. A pesar de su frecuente utilización presenta un estrecho rango terapéutico que justifica conocer 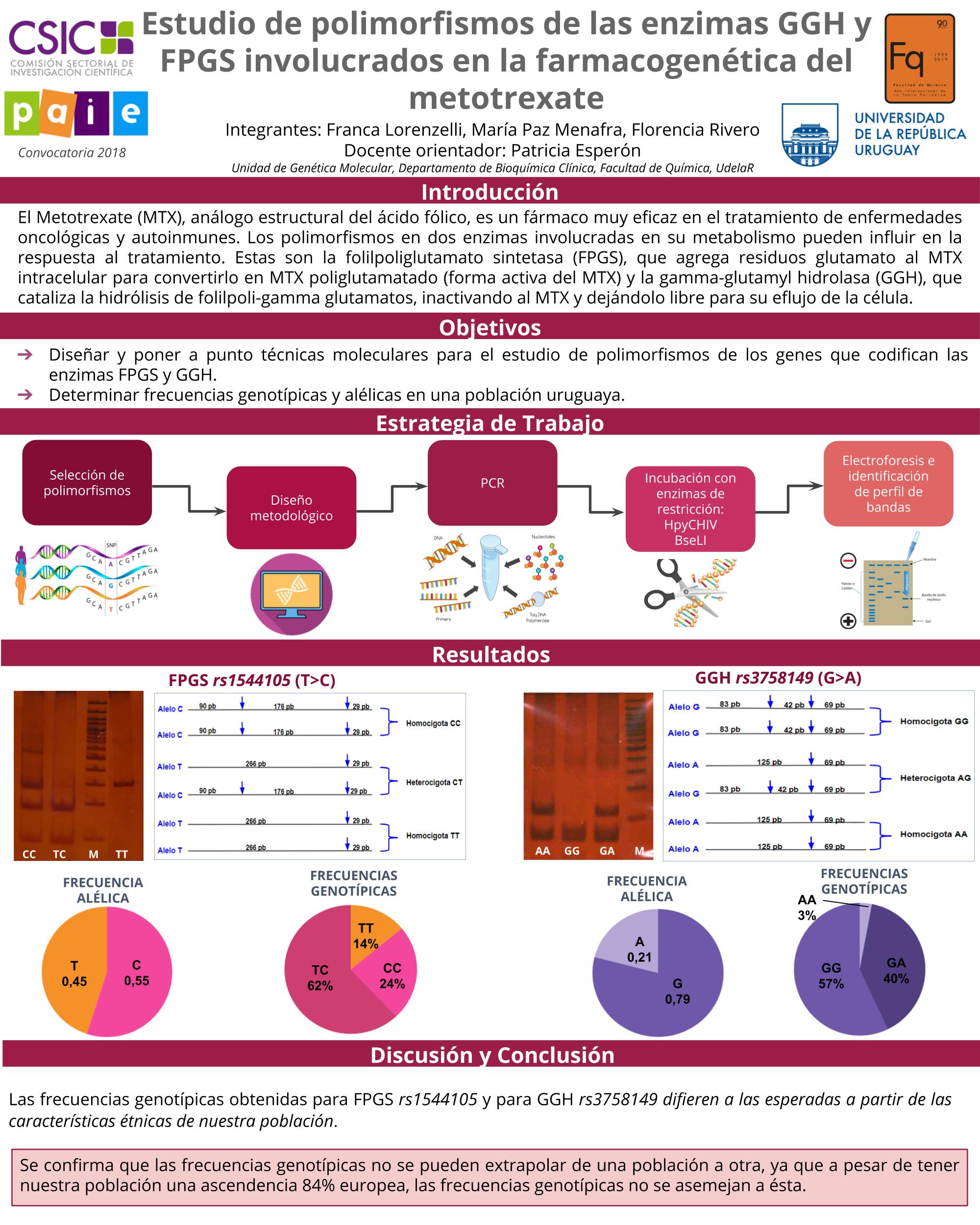 diferentes parámetros en el ajuste de dosis. Dos de las enzimas involucradas en el metabolismo del MTX son la folilpoliglutamato sintetasa (FPGS), y la gamma-glutamyl hidrolasa (GGH), las cuales actúan activando e inactivando el MTX respectivamente. En este trabajo se plantea el diseño de técnicas moleculares para el estudio de polimorfismos de estas enzimas en una población uruguaya. La elección de los polimorfismos se apoyó en una búsqueda bibliográfica, seleccionando aquellos para los cuales se reportó una asociación con la eficacia y/o toxicidad en el tratamiento con MTX. Se seleccionaron las variantes FPGS rs1544105 y GGH rs3758149. Se realizó la extracción de ADN a partir de sangre periférica empleando un kit comercial. Se realizó el diseño de los primers en base a las secuencias obtenidas de Ensembl genome browser 95 utilizando los programas Gene Runner, Primer3 y Oligoanalyzer. Se utilizó como método la Reacción en Cadena de la Polimerasa en su variante “Polimorfismo en la Longitud de los Fragmentos de Restricción” (PCR-RFLP). Se pusieron a punto las condiciones de PCR, ensayando distintas concentraciones de reactivos y programas de ciclado. Se seleccionaron dos enzimas de restricción, una para cada polimorfismo, y se ajustaron las condiciones de temperatura y tiempos de incubación. Se amplificó cada muestra y se incubaron con las enzimas de restricción. Se realizaron dos electroforesis en gel de poliacrilamida, una para corroborar la correcta amplificación del producto de PCR, y otra luego de la incubación, donde se observaron distintos perfiles de bandas que se relacionaron por los tamaños esperados, con los correspondientes genotipos. Como método de confirmación se amplificaron, purificaron y secuenciaron dos ADNs distintos para cada polimorfismo. Con los datos obtenidos se calcularon las frecuencias genotípicas: para FPGS 62% TC, 24% CC y 14% TT; y para GGH 40% GA, 57% GG y 3% AA. Este trabajo logra poner a punto una técnica molecular para el estudio de dos polimorfismos de las enzimas FPGS y GGH, hasta entonces no estudiados en el Uruguay.
diferentes parámetros en el ajuste de dosis. Dos de las enzimas involucradas en el metabolismo del MTX son la folilpoliglutamato sintetasa (FPGS), y la gamma-glutamyl hidrolasa (GGH), las cuales actúan activando e inactivando el MTX respectivamente. En este trabajo se plantea el diseño de técnicas moleculares para el estudio de polimorfismos de estas enzimas en una población uruguaya. La elección de los polimorfismos se apoyó en una búsqueda bibliográfica, seleccionando aquellos para los cuales se reportó una asociación con la eficacia y/o toxicidad en el tratamiento con MTX. Se seleccionaron las variantes FPGS rs1544105 y GGH rs3758149. Se realizó la extracción de ADN a partir de sangre periférica empleando un kit comercial. Se realizó el diseño de los primers en base a las secuencias obtenidas de Ensembl genome browser 95 utilizando los programas Gene Runner, Primer3 y Oligoanalyzer. Se utilizó como método la Reacción en Cadena de la Polimerasa en su variante “Polimorfismo en la Longitud de los Fragmentos de Restricción” (PCR-RFLP). Se pusieron a punto las condiciones de PCR, ensayando distintas concentraciones de reactivos y programas de ciclado. Se seleccionaron dos enzimas de restricción, una para cada polimorfismo, y se ajustaron las condiciones de temperatura y tiempos de incubación. Se amplificó cada muestra y se incubaron con las enzimas de restricción. Se realizaron dos electroforesis en gel de poliacrilamida, una para corroborar la correcta amplificación del producto de PCR, y otra luego de la incubación, donde se observaron distintos perfiles de bandas que se relacionaron por los tamaños esperados, con los correspondientes genotipos. Como método de confirmación se amplificaron, purificaron y secuenciaron dos ADNs distintos para cada polimorfismo. Con los datos obtenidos se calcularon las frecuencias genotípicas: para FPGS 62% TC, 24% CC y 14% TT; y para GGH 40% GA, 57% GG y 3% AA. Este trabajo logra poner a punto una técnica molecular para el estudio de dos polimorfismos de las enzimas FPGS y GGH, hasta entonces no estudiados en el Uruguay.
María Paz Menafra; Florencia Rivero; Patricia Esperón Unidad de Genética Molecular, Departamento de Bioquímica Clínica, Facultad de Química, UdelaR



