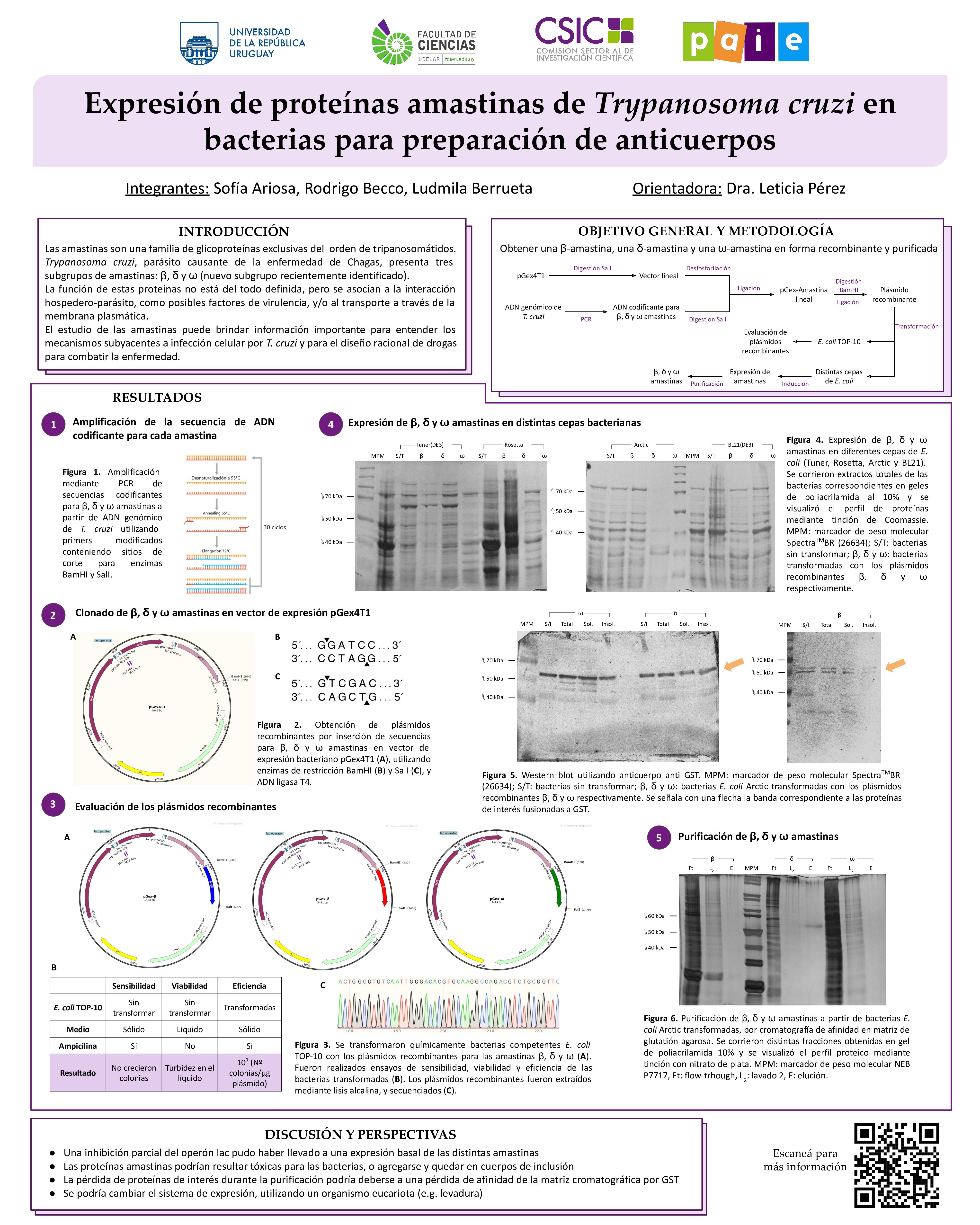
El proyecto “Expresión de proteínas amastinas de Trypanosoma cruzi en bacterias para preparación de anticuerpos” realizado por Sofía Ariosa, Rodrigo Becco y Ludmila Berrueta, orientado por la Dra. Leticia Pérez en el Laboratorio de Interacciones Moleculares de Facultad de Ciencias, consistió en la obtención de proteínas amastinas (β, δ y ω) en su forma recombinante y purificada. Se generó ADN recombinante insertando la secuencia codificante para las amastinas en un vector de expresión bacteriano, utilizando enzimas de restricción para generar extremos de unión cohesivos entre el vector y el inserto. Este ADN se incorporó a bacterias E. coli, y posteriormente fue extraído de las mismas para ser secuenciado y servir como verificación de los pasos experimentales anteriores. Una vez confirmada la correcta secuencia del plásmido recombinante, se transformaron bacterias E. coli Arctic, BL21(DE3), Tuner(DE3) y Rosetta, a las que se les indujo la expresión de las proteínas. Las condiciones de expresión se pusieron a punto modificando temperatura y tiempo de inducción con IPTG. La expresión de amastinas fue confirmada mediante Western-blot, con anticuerpo anti GST. Finalmente, se obtuvieron las fracciones celulares de los cultivos, se realizó la purificación de las amastinas mediante cromatografía de afinidad utilizando una matriz de glutatión agarosa, procedimiento que fue evaluado a través de electroforesis en geles de poliacrilamida. En este proyecto se obtuvieron bacterias que expresan β, δ y ω amastinas, pero solo pudo aislarse δ amastina (β y ω co-purificaban con otras proteínas o se perdían en el proceso de purificación).